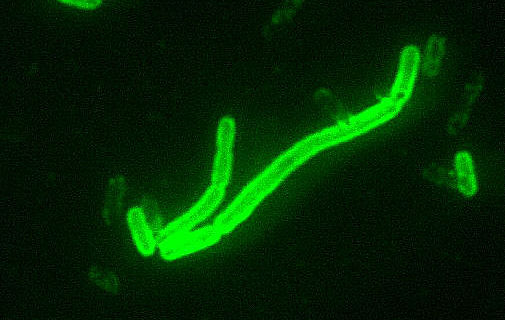
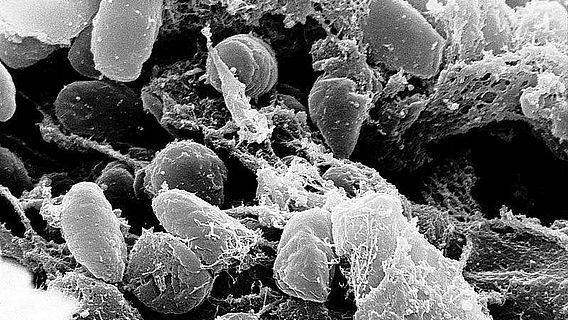
Genom des mittelalterlichen Lepraerregers rekonstruiert
Unter der Leitung von Prof. Johannes Krause von der Universität Tübingen und Prof. Stewart Cole von der Eidgenössischen Technischen Hochschule Lausanne (EPFL) erstellte die Forschungsgruppe erstmals ein Genom aus archäologischen Funden, ohne auf eine Referenzsequenz zurückzugreifen.
Möglich war dies durch eine außergewöhnlich gut erhaltene DNA der mittelalterlichen Krankheitserreger. Die Ergebnisse deuten darauf hin, dass alle nordamerikanischen Lepra-Stämme, die heute in Gürteltieren und Menschen gefunden werden, europäischen Ursprungs sind und auf einen gemeinsamen Vorfahren zurückgehen, der vor weniger als 4000 Jahren lebte. Zudem lässt der Befund vermuten, dass Bakterien-DNA weit länger erhalten bleiben kann als die DNA von Wirbeltieren, die schätzungsweise rund eine Million Jahre nachweisbar bleibt.
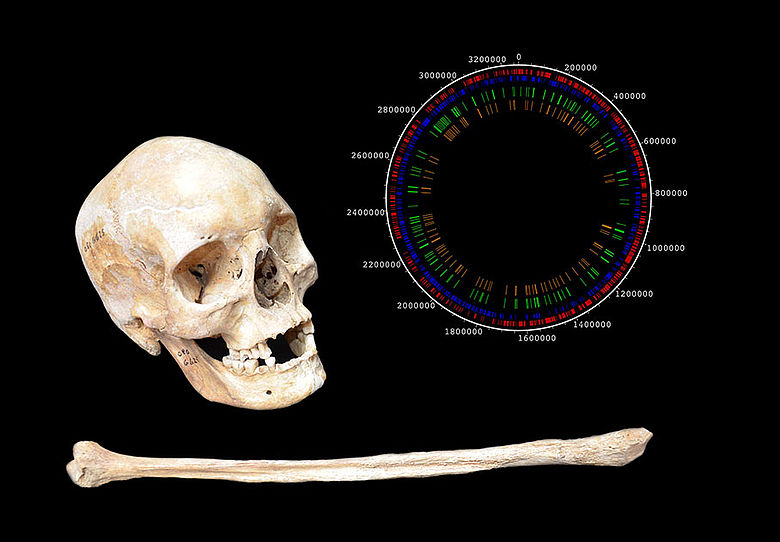
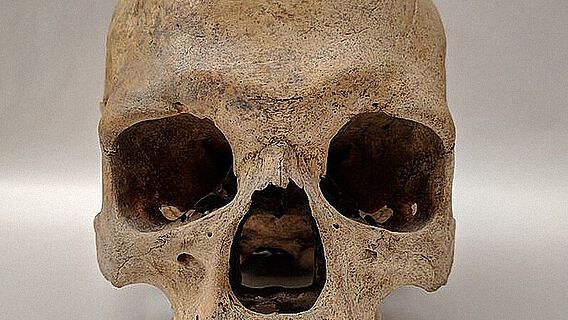
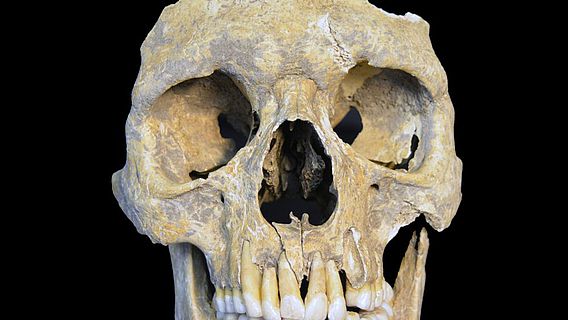
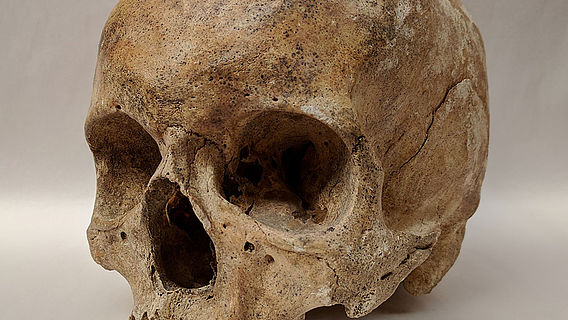
Lepra, eine verheerende ansteckende, chronische Krankheit, war bis ins späte Mittelalter in Europa weit verbreitet. Die Erkrankten wurden in eigens errichteten Leprakolonien isoliert. Heute ist die Krankheit weltweit in 91 Ländern zu finden, mit mehr als 200.000 neuen Infektionen jährlich. Um die Geschichte der Krankheit zurückzuverfolgen, hatten die Wissenschaftler die kompletten Genome von M. leprae Bakterien aus fünf mittelalterlichen Skeletten aus Dänemark, Schweden und Großbritannien rekonstruiert, welche die für Lepra charakteristischen Knochenveränderungen aufwiesen. Zusätzlich wurde die M. leprae Erbsubstanz aus sieben Biopsie-Proben heutiger Patienten entschlüsselt.
Die Forscher verglichen die mittelalterlichen M. leprae Genome aus Europa mit denen der sieben Biopsien und vier weiterer moderner Bakterienstämme. Sie stellten fest, dass alle M. leprae Stämme einen gemeinsamen Vorfahren haben, der vor weniger als 4000 Jahren existierte. Dieses Ergebnis wird unterstützt durch den frühesten archäologischen Beleg für die Krankheit in Indien. Die genomweiten Vergleiche deuten auf eine außergewöhnlich geringe Veränderung der Erbsubstanz der Bakterien innerhalb der letzten 1000 Jahre hin, die höchst wahrscheinlich keine Auswirkungen auf die Virulenz der Erreger hatte. Dieser Befund legt nahe, dass das Ende der Lepra-Epidemie im Mittelalter durch andere Faktoren wie beispielsweise verbesserte soziale Verhältnisse beeinflusst worden ist. Das Wissenschaftlerteam konnte zudem zeigen, dass eine M. leprae Form, die im Mittelalter in Europa vorkam, aktuell im mittleren Osten beobachtet wird. Ein anderer mittelalterlicher Stamm aus Europa weist frappierende Ähnlichkeiten auf zu Bakterien, die heute in Gürteltieren und Lepra-Patienten in Nordamerika vorkommen, was auf einen europäischen Ursprung der Erkrankung in Amerika deutet.
Überraschenderweise war in den untersuchten Skeletten eine viel größere Menge Erreger-DNA vorhanden, als normalerweise in heutigen Patienten zu finden ist. Die Autoren führen dieses Phänomen darauf zurück, dass sich bakterielle DNA wahrscheinlich auf Grund der extrem dicken Zellwand des Lepra-Bakteriums nur sehr langsam zersetzt und sich so in den Skeletten über die Jahre anreichert. »Das eröffnet die Möglichkeit, dass bestimmte Formen bakterieller DNA über das maximale Alter für Säugetier-DNA hinaus erhalten bleibt, das rund eine Million Jahre beträgt«, sagt Johannes Krause. »Damit sollte es möglich sein, die Krankheit bis in ihre prähistorischen Ursprünge zurückzuverfolgen.«
»Vor allem in einem Skelett aus Dänemark war die DNA des Krankheitserregers außergewöhnlich gut erhalten, obwohl die Erkrankung bei der etwa 25 Jahre alten Frau gar nicht so stark ausgeprägt war«, sagt Prof. Almut Nebel von der Universität Kiel. Diese DNA-Probe ermöglichte es zum ersten Mal, ein Genom eines historischen Krankheitserregers von Grund auf neu, ohne Vergleich mit einer Referenzsequenz zusammenzusetzen. »Wir haben den Kontakt zu den dänischen Kollegen hergestellt, welche die für diese Studie wichtigen Skelette zur Verfügung gestellt haben. Aus den Zähnen der Leprakranken haben wir die DNA gewonnen und diese einer ersten molekularen Eignungsprüfung unterzogen«, berichtet Ben Krause-Kyora. Außerdem wiederholten die Kieler Untersuchungen der Tübinger Kollegen, um die außergewöhnlichen Ergebnisse unabhängig zu belegen. Das Kieler Forschungsprojekt wurde von der Graduiertenschule Human Development in Landscapes und dem Exzellenzcluster Entzündungsforschung unterstützt.
Publikation
The history of leprosy from genome-wide comparison of medieval and modern Mycobacterium leprae, Verena J. Schuenemann, Pushpendra Singh, Thomas A. Mendum, Ben Krause-Kyora, Günther Jäger, et al., Science
DOI: 10.1126/science.1238286
Online publiziert am 13.06.2013
Folgende Autoren haben zu dieser Publikation beigetragen:
Verena J. Schuenemann, Günter Jäger, Kirsten I. Bos, Alexander Herbig, Peter Bauer, Kay Nieselt und Johannes Krause von der Universität Tübingen (Deutschland); Pushpendra Singh, Andrej Benjak, Philippe Busso und Stewart T. Cole von der Ecole Polytechnique Fédérale de Lausanne (Schweiz); Thomas A. Mendum, Huihai Wu, Graham R. Stewart and G. Michael Taylor von der Universität Surrey (Großbritannien); Ben Krause-Kyora und Almut Nebel von der Universität Kiel (Deutschland); Christos Economou und Anna Kjellström von Universität Stockholm (Schweden); Jesper L. Boldsen von der Medicine University of Southern Denmark (Dänemark); Oona Y.-C. Lee, Houdini H.T. Wu, David E. Minnikin and Gurdyal S. Besra von der Universität Birmingham (Großbritannien); Katie Tucker und Simon Roffey von der Universität Winchester (Großbritannien); Samba O. Sow vom Zentrum für Impfstoff Entwicklung-Mali (Mali).
RSS-Feeds @ Archäologie Online
- Nachrichten
- Videos
- Podcasts