Die Genome der Opfer des Schwarzen Todes entschlüsseln
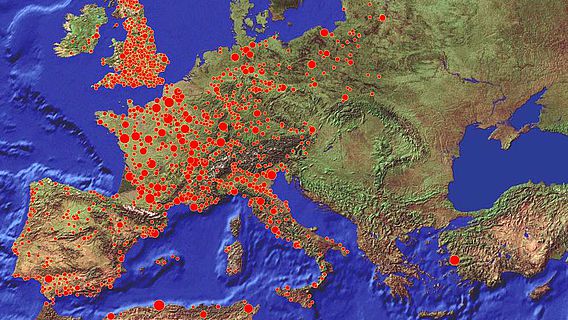
In den letzten zehn Jahren haben Forscherinnen und Forscher die genetischen Daten von mehr als 10.000 Menschen aus der Vergangenheit untersucht. Diese Datenfülle hat zu völlig neuen Erkenntnissen darüber geführt, wie sich große Menschengruppen in der Vergangenheit vermischt und im Laufe der Zeit verändert haben und wie bestimmte genetische Merkmale gegenüber anderen selektiert wurden. Die Untersuchung jüngerer genetischer Veränderungen in Populationen, die sich ständig in Bewegung befanden, wie dies im mittelalterlichen und modernen Europa der Fall war, blieb jedoch wegen methodischer Einschränkungen und einem begrenzten Zugang zu historischer alter DNA eine Herausforderung.
"Um besser zu verstehen, warum bestimmte Krankheiten in einigen modernen Gruppen häufiger auftreten als in anderen, und um Veränderungen in bestimmten Genen mit den Faktoren in Verbindung bringen zu können, die zu diesen Veränderungen geführt haben, müssen wir unser Wissen über die jüngere Populationsgeschichte des Menschen ausbauen", sagt Projektleiter Harald Ringbauer. Besonders deutlich wird diese Lücke bei der Erforschung des Schwarzen Todes, der tödlichen ersten Welle der mittelalterlichen Pest, die im 14. Jahrhundert Millionen Menschen in Europa dahinraffte. Zwar gibt es Hinweise darauf, dass der Schwarze Tod dauerhafte Veränderungen in den Genen unseres Immunsystems ausgelöst hat, die auch heute noch von Bedeutung sind. Allerdings sind sich die Experten nicht einig, wie die spärlichen Daten zu interpretieren sind, so dass ein genaues Verständnis der generellen Auswirkungen der Pest auf das menschliche Genom bisher nicht möglich war.
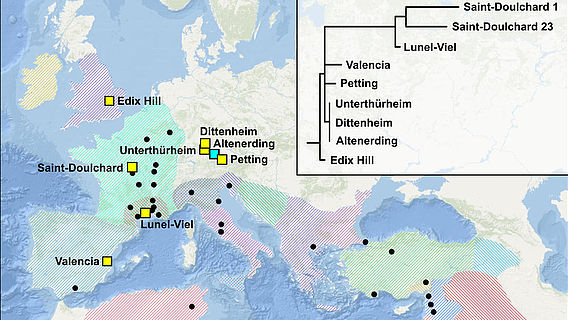
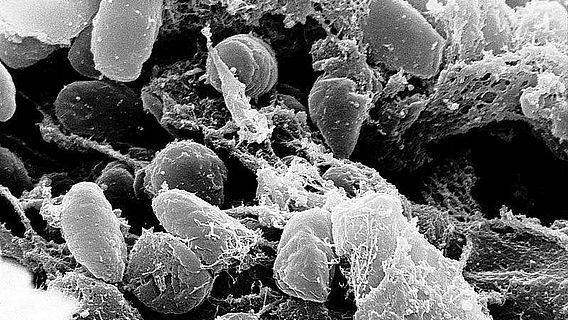
Um diese Lücken zu schließen, werden Ringbauer und sein Team die Genome von 500 Opfern des Schwarzen Todes aus St. Pölten, Österreich, sequenzieren und analysieren. Die Ausgrabungen auf dem Domplatz der Stadt dauerten fast ein Jahrzehnt und brachten einen riesigen mittelalterlichen Friedhof mit fast 22.500 Skeletten ans Licht. Mehr als 500 von ihnen wurden in drei erstaunlichen Massengräbern beigesetzt. Forscher des MPI-EVA haben nun die Todesursache aufgedeckt: In den menschlichen Überresten identifizierten sie DNA von Yersinia pestis, die exakt mit dem Stamm übereinstimmt, der die erste Pestwelle auslöste. "Unser erstes großes Ziel ist es, die einmalige Chance zu nutzen, die uns die große Zahl der jetzt identifizierten Opfer des Schwarzen Todes bietet, um besser zu verstehen, in welcher Beziehung diese Menschen, die alle im Jahr 1350 starben, zu den heute in Europa lebenden Menschen stehen", sagt Ringbauer. "Neue computergestützte Methoden werden es uns ermöglichen, in die Vergangenheit zu reisen und diese Generation mittelalterlicher Menschen mit den heutigen Europäern in Verbindung zu bringen und die Bevölkerungsstruktur und Demografie über die Jahrhunderte hinweg zu untersuchen."
Das zweite große Ziel des Teams ist es, anhand der Genome der Opfer des Schwarzen Todes spezifische genetische Merkmale zu identifizieren, die mit einer erhöhten Anfälligkeit oder Resistenz gegen die Krankheit einhergehen. "Genetische Varianten, die das Risiko eines tödlichen Ausgangs einer Infektion mit dem Schwarzen Tod erhöhen, werden in den Genomen der 500 Opfer gehäuft auftreten. Die große Stichprobengröße ermöglicht es uns, diese Varianten zu finden und erstmals die direkten Auswirkungen des Schwarzen Todes auf die menschliche Genetik gründlich zu untersuchen und möglicherweise wichtige genetische Informationen zu entdecken, die für aktuelle und zukünftige Krankheitsausbrüche von Bedeutung sind", so Ringbauer weiter. Um dieses Ziel zu erreichen, wird das Genomforschungsteam weiterhin eng mit dem St. Pöltner Stadtarchäologen Ronald Risy, der die Ausgrabungen leitete, und dem Anthropologen und Professor an der Medizinischen Universität Wien, Fabian Kanz, zusammenarbeiten, dessen Team aus der Analyse der Skelette relevante Kontextdaten für die Opfer des Schwarzen Todes - wie Alter und Ernährungszustand - erstellen wird.
RSS-Feeds @ Archäologie Online
- Nachrichten
- Videos
- Podcasts