Einem internationalen Team unter der Leitung von Wissenschaftlerinnen und Wissenschaftlern der Christian-Albrechts-Universität zu Kiel (CAU) im Rahmen des Sonderforschungsbereiches 1266 »TransformationsDimensionen« und des Max-Planck-Instituts für Menschheitsgeschichte (MPI) Jena gelang die Rekonstruktion von Genomen steinzeitlicher und mittelalterlicher Stämme des Hepatitis-B-Virus (HBV). Hepatitis B zirkulierte bereits seit ca. 7.000 Jahren in Europa, und obwohl diese neu entdeckten HBV-Varianten denen heutiger Stämme ähnlich sind, stellen sie früher eine eigene, mittlerweile vermutlich ausgestorbene Abstammungslinie dar. Die nächsten Verwandten kommen heute in Schimpansen und Gorillas vor.
Mit weltweit über 250 Millionen infizierten Menschen zählt der Hepatitis-B-Virus heutzutage zu den am weitesten verbreiteten bekannten Krankheitserregern. Geschichte und Epidemiologie in prähistorischen Populationen waren hingegen noch weitestgehend unbekannt. Bisher war die Extrahierung der viralen aDNA (ancient DNA) aus archäologischen Proben noch nicht gelungen – Herkunft und Evolution des Virus blieben den Forschern unzugänglich.
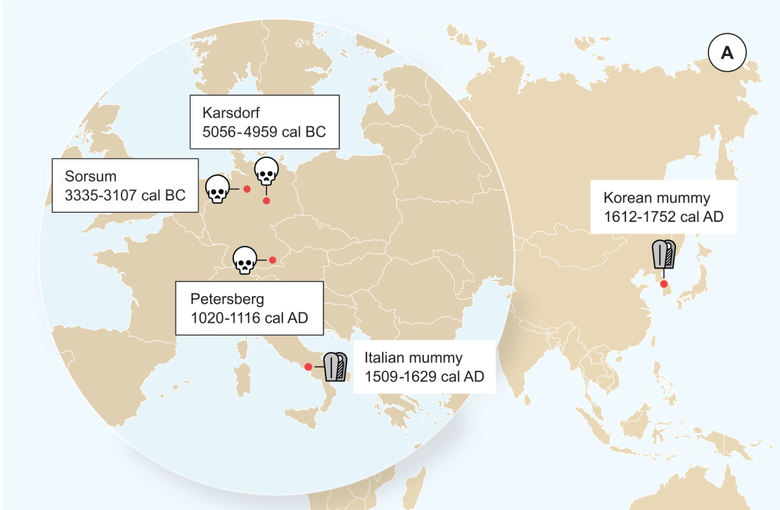
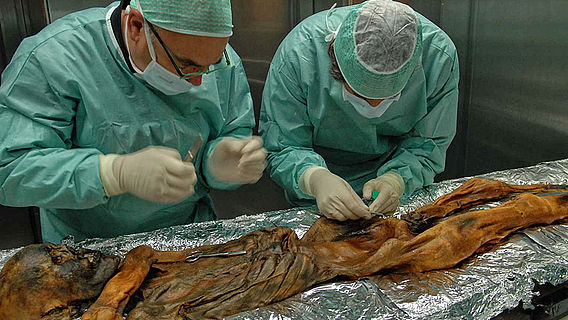
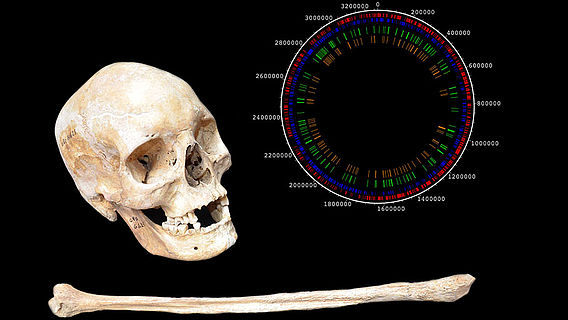
In einer aktuellen Studie gelang es dem internationalen Forschungsteam vom Institut für Klinische Molekularbiologie (IKMB) der CAU und dem MPI Jena erstmals die Gewinnung viraler aDNA aus Zahnproben zweier jungsteinzeitlicher Individuen und eines mittelalterlichen Individuums. Das ermöglichte die Rekonstruktion dreier HBV-Stämme. Der Befund aus Karsdorf, Sachsen-Anhalt, stellt mit ca. 7.000 Jahren den ältesten genetisch nachgewiesenen viralen Krankheitserreger dar. Die Erkenntnisse werden am Donnerstag, 10. Mai, in dem Fachjournal »eLIFE« veröffentlicht.
Die komplexe Evolution von Hepatitis B
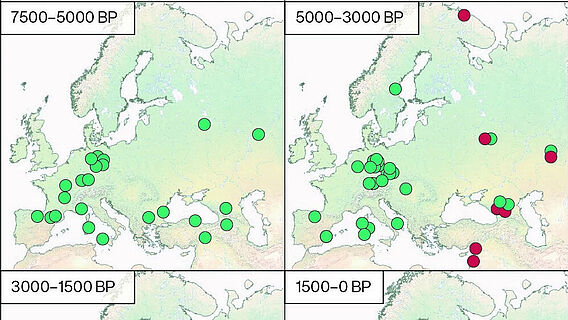
Für die Studie wurden 53 Skelette aus neolithischen und mittelalterlichen Fundstätten in Deutschland beprobt, die insgesamt einen Zeitraum von 5.000 vor bis 1.200 nach Beginn unserer Zeitrechnung umspannen. Mittels modernster Genanalysetechniken wurden Proben auf die Präsenz viraler Pathogene überprüft, wobei das Forschungsteam bei drei Individuen alte Hepatitis-B-Viren nachweisen und deren Genome rekonstruieren konnten. Diese Virus-Genome scheinen eigene Abstammungslinien darzustellen, die keine nahe Verwandtschaft zu heutigen menschlichen Stämmen aufweisen und daher vermutlich ausgestorben sind, vermuten die Forscherinnen und Forscher.
Im Vergleich zum mittelalterlichen sind sich die zwei neolithischen Genome relativ ähnlich, wobei deren Träger in einem Abstand von ca. 2.000 Jahren lebten. Die steinzeitlichen Erreger ähneln am ehesten denen in heute vorkommenden HBV bei nicht-menschlichen Primaten wie Schimpanse und Gorilla. Im Gegensatz dazu ist das mittelalterliche Genom den heutigen Stämmen ähnlicher, stellt aber dennoch eine eigene Linie dar. Dies gilt selbst für den Vergleich mit zwei bereits bekannten historischen HBV-Genomen, die aus Mumien des 16. Jahrhunderts gewonnen werden konnten. Diese HBV-Stämme sind eng verwandt mit modernen Varianten, was auf erstaunlich geringe Evolution des Virus in den letzten 500 Jahren hinweist. Insgesamt lassen die Ergebnisse auf eine komplexe Geschichte von Mehrfachübertragungen zwischen Mensch und nicht-menschlichen Primaten schließen.
»Alles in allem zeigen unsere Ergebnisse, dass das Hepatitis-B-Virus bereits vor 7.000 Jahren bei Europäern vorkam. Zudem ist seine Genomstruktur der des heutigen Hepatitis-B-Virus sehr ähnlich, ungeachtet der beobachteten Unterschiede«, so der Erstautor der Studie Ben Krause-Kyora, Professor am IKMB der Universität Kiel und Wissenschaftler im SFB 1266. »Es müssen sowohl ältere Vorläufer des Hepatitis-B-Virus als auch mehr heutige Virusstämme sowie ihre zwischenzeitlichen Entwicklungsformen untersucht werden, um die komplexe Evolution von Hepatitis zu entwirren.«
Neue Analysemethoden zur Erforschung blutübertragbarer Viren
Almut Nebel, Wissenschaftlerin am IKMB und Co-Autorin der Studie, erklärt den größeren Zusammenhang: »Wir untersuchen, ob es einen Zusammenhang zwischen dem Aufkommen von Krankheiten und grundlegenden Veränderungen in der Lebensweise der Menschen in der Ur- und Frühgeschichte gibt. Dank moderner Methoden sind wir in der Lage, diese Zusammenhänge zu entschlüsseln, indem wir die menschliche und die pathogene Genetik der damaligen Zeit untersuchen.« Gemeinsam mit Krause-Kyora leitet sie Teilprojekt F4 im Sonderforschungsbereich 1266 »TransformationsDimensionen« der Kieler Universität, welches die Rolle von Infektionskrankheiten im Hinblick auf demographischen Wandel und die Beziehung von Menschen und ihrer Umwelt im Zeitraum 15.000 bis zu Beginn unserer Zeitrechnung untersucht. Die Individuen vom neolithischen Fundplatz Sorsum und dem mittelalterlichen Grabkontext Petersberg waren Teil einer großangelegten Studie des Teilprojektes, um virale und bakterielle Pathogene an verlässlich datierten Skeletten dieser Zeitspanne zu erfassen.
Begeistert zeigt sich Krause-Kyora über die Möglichkeiten moderner aDNA-Forschung und den Möglichkeiten wissenschaftlichen Vernetzung: »Seit wir an der CAU mit der Erforschung alter menschlicher und pathogener DNA begonnen haben, hat sich viel getan. Junge Disziplinen wie die Proteomik erweitern das Methodenspektrum um alte Krankheiten und das menschliche Genom vor einem archäologischen und medizinischen Hintergrund zu untersuchen. In der Zusammenarbeit mit den Wissenschaftlern des MPI haben wir eine nachhaltige Struktur in diesem vergleichsweise jungen Forschungsfeld geschaffen, um gemeinsam komplexe Fragestellungen mit aktuellem Bezug zu untersuchen.«
Johannes Krause, Seniorautor und Direktor der Abteilung Archäogenetik am MPI für Menschheitsgeschichte, betont die Bedeutung der Studie. »Unsere Ergebnisse zeigen das große Potenzial von aus menschlichen Knochen gewonnener aDNA. Sie erlaubt uns, die Evolution von durch Blut übertragenen Viren zu erforschen. Bisher war es immer zweifelhaft, ob dies möglich wäre und überhaupt solche Erkrankungen für vergangene Zeiten nachzuweisen. Nun haben wir ein leistungsfähiges Mittel gefunden, um die vielschichtige Evolutionsgeschichte von Viruserkrankungen zu erkunden.«
Publikation
Neolithic and Medieval virus genomes reveal complex evolution of Hepatitis B
eLife. 10.05.2018
DOI: 10.7554/eLife.36666