Wenn zwei Personen biologisch miteinander verwandt sind, teilen sie lange Abschnitte der DNA, die sie von ihrem letzten gemeinsamen Vorfahren geerbt haben. Diese beinahe identischen Genomabschnitte werden als IBD-Segmente (»Identity by Descent«) bezeichnet. Bis zum sechsten Grad der Verwandtschaft – bis hin zu Cousins und Cousinen zweiten oder dritten Grades oder einem Ururururgroßelternteil – haben beide Verwandte sogar mehrere IBD-Segmente gemein. Unternehmen wie 23andme oder Ancestry erkennen diese Segmente routinemäßig im Erbgut ihrer Kunden und nutzen dieses Signal, um biologische Verwandte in ihren Datenbanken eindeutig zu identifizieren.
In einer neuen Studie haben Forschende des Leipziger Max-Planck-Instituts für evolutionäre Anthropologie und der Harvard University in den USA nun ein leistungsfähiges neues Instrument namens »ancIBD« entwickelt, um diese IBD-Segmente auch in Genomen von Menschen aufzuspüren, die vor Hunderten, Tausenden oder sogar Zehntausenden von Jahren gelebt haben. Um der Herausforderung zu begegnen, dass solche alten Genome oft sehr stark degradiert und somit im Vergleich zu moderner DNA von sehr viel schlechterer Qualität sind, mussten die Autoren in die innovative »Trickkiste« greifen und Lücken in alten Genomen mit moderner Referenz-DNA auffüllen.
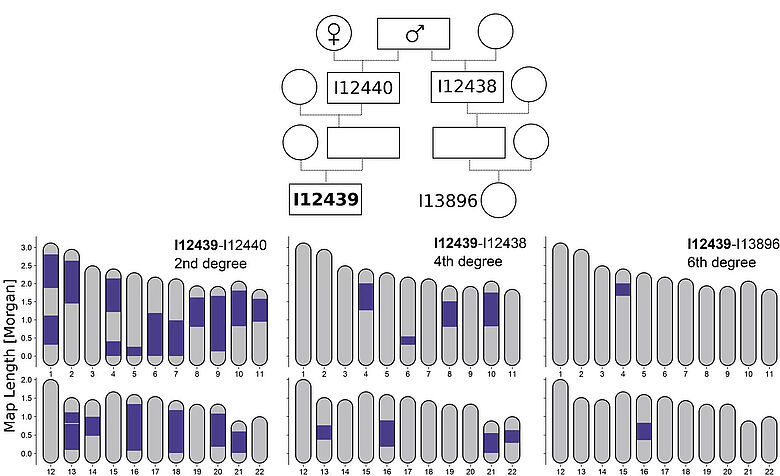
Diese Innovation eröffnet den Forschenden nun völlig neue Möglichkeiten, alte DNA-Daten zu analysieren. »Indem wir Regionen im Genom, die Individuen miteinander gemein haben, genauestens bestimmen, können wir nun auch in alten Genomen Paare von Verwandten bis hin zum sechsten Grad aufspüren, während frühere Methoden, die auf der Bestimmung genomischer Durchschnittsähnlichkeiten basierten, nur Verwandte bis zum dritten Grad entdecken konnten«, erklärt Yilei Huang, ein Erstautor der Studie und Doktorand am Max-Planck-Institut für evolutionäre Anthropologie.
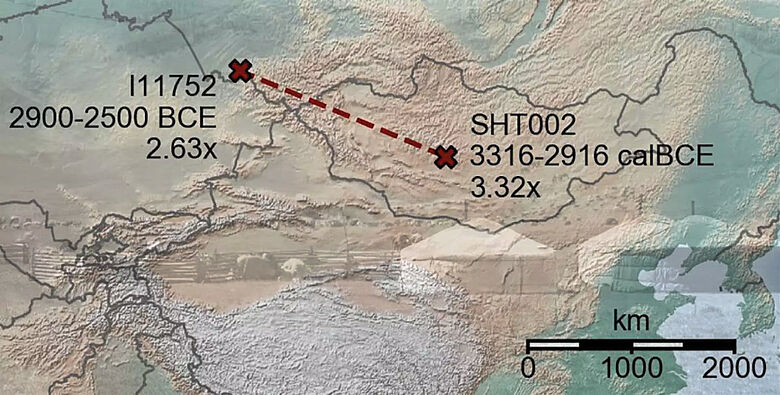
Mit der neuen Methode untersuchten die Autoren einen Datensatz von 4.248 bereits veröffentlichten alten Genomen aus ganz Eurasien aus den letzten 50.000 Jahren und konnten Hunderte von zuvor unentdeckten Verwandtenpaaren identifizieren. In einigen besonders faszinierenden Fällen waren beide Verwandten sehr weit entfernt voneinander bestattet – ein direkter Nachweis für die hohe Mobilität dieses Verwandtschaftsclusters. In einem solchen Fall entdeckten die Autoren ein Paar von zwei frühbronzezeitlichen Nomaden aus Zentralasien, die vor ca. 5.000 Jahren lebten und Verwandte fünften Grades waren – ihre jeweiligen Gräber waren ca. 1.500 Kilometer voneinander entfernt! Diese Menschen oder ihre unmittelbaren Vorfahren müssen zwischen ihrer Geburt und ihrer Bestattung Hunderte von Kilometern zurückgelegt haben.
Das neue Analysetool ermöglicht es den Autoren, Verwandtschaftsbeziehungen sogar noch viel weiter zurückzuverfolgen und mit einer noch nie dagewesenen Präzision zu untersuchen. Nicht alle Verwandten jenseits des zehnten Grades teilen lange IBD-Segmente. Dennoch ist es den Autoren gelungen, anhand der Bestimmung einer durchschnittlichen Rate identischer langer DNA-Segmente bisher unbekannte Verbindungen zwischen Gruppen prähistorischer und historischer Menschen nachzuweisen. »Wir haben spannende neue Verbindungen zwischen alten Kulturen aufgespürt – und anhand des Signals, den langen identischen DNA-Segmenten, ist es uns nun auch erstmals gelungen, enge Beziehungen zwischen bedeutsamen alten Kulturen nachzuweisen, manchmal über weite Entfernungen und einen Zeitraum von nur wenigen hundert Jahren hinweg«, sagt Harald Ringbauer vom Max-Planck-Institut für evolutionäre Anthropologie, der diese Studie leitete.
Genfluss aus der eurasischen Steppe
So enthüllten die Autoren unter anderem faszinierende neue Details über einen massiven Genfluss aus der eurasischen Steppe, der vor ca. 5.000 Jahren begann. Die ersten Europäer mit substantiellem Anteil an Steppenabstammung, die mit der Schnurkeramik assoziiert werden, einer archäologischen Kultur, die sich damals von Mitteleuropa bis Skandinavien und das heutige Russland ausbreitete, teilen sich viele lange IBD-Segmente mit den Jamnaja-Hirten der pontisch-kaspischen Steppe. Das deutet auf ein ausgeprägtes genetisches Flaschenhals-Ereignis und eine nur wenige hundert Jahre zurückliegende biologische Verbindung zwischen diesen Gruppen von Populationen hin.
Außerdem spürten die Autoren lange IBD-Segmente auf, die sich Gruppen von Menschen der Schnurkeramik-Kultur mit Osteuropäern teilten, die mit der Kugelamphoren-Kultur aus Polen und der Ukraine assoziiert waren und noch keine Steppenabstammung aufwiesen. »Diese IBD-Verbindungen finden wir bei allen Schnurkeramik-Gruppen von Zentraleuropa bis Russland. Menschen mit Kugelamphoren-Kontext scheinen also schon sehr frühzeitig in der Entstehung verschiedener Schnurkeramik-Gruppen einen bedeutenden demographischen Einfluss gehabt zu haben; es kam zu genetischen Vermischungen,« sagt Ringbauer.
Die neue Methode zum Screening alter DNA auf elterliche Verwandtschaft gibt Wissenschaftlern ein vielseitig einsetzbares bioinformatisches Instrument an die Hand. Auch zukünftig wird die Forschung auf dem Gebiet der alten DNA rasant wachsen – mit Tausenden neu verfügbaren Genomen pro Jahr. So können Forschende das Leben unserer Vorfahren weiter erkunden – auf Mikroebene, mit Fokus auf individuellen Lebenswegen damals lebender Menschen und ihrer Verwandten, und auf Makroebene, wenn es darum geht, bedeutende kulturhistorische Ereignisse in neuer Detailtiefe zu erforschen.
Publikation
Accurate detection of identity-by-descent segments in human ancient DNA
Nature Genetics. 20.12.2023
DOI: 10.1038/s41588-023-01582-w
https://www.nature.com/articles/s41588-0...